Diferencia entre enlazador y adaptador
El diferencia clave Entre vinculador y adaptador es que Un enlazador no tiene extremos cohesivos, mientras que un adaptador tiene un extremo cohesivo.
La ligadura de ADN es el proceso de unir dos moléculas de ADN juntas, formando enlaces fosfodiesteros. La enzima llamada ADN ligasa cataliza esta reacción. Es uno de los pasos críticos en los campos biológicos moleculares modernos, como la tecnología de ADN recombinante y la clonación de ADN. La eficiencia de ligadura depende de los extremos de las moléculas de ADN a ser ligadas. Hay dos tipos de extremos de ADN como extremos pegajosos y extremos contundentes. La eficiencia de la ligadura es alta con los extremos pegajosos que con los extremos contundentes. Si las moléculas de ADN objetivo tienen extremos contundentes, las moléculas llamadas adaptadores o enlazadores serán útiles. Los adaptadores y los enlazadores son moléculas de oligonucleótidos sintetizadas químicamente que ayudan en la ligadura de ADN. Tienen sitios de restricción interna también. El adaptador tiene un extremo adhesivo y un extremo contundente, mientras que el enlazador tiene dos extremos contundentes.
CONTENIDO
1. Descripción general y diferencia de claves
2. ¿Qué es un enlazador?
3. ¿Qué es un adaptador?
4. Similitudes entre enlazador y adaptador
5. Comparación de lado a lado: adaptador VS en forma tabular
6. Resumen
¿Qué es un enlazador??
Linker es una secuencia de oligonucleótidos sintetizada químicamente que es doble. Linker tiene dos extremos contundentes. El enlazador se usa para ligar moléculas de ADN que tienen extremos rectos a los vectores. Contiene uno o más sitios de restricción interna. Estos sitios de restricción funcionan como sitios de reconocimiento para enzimas de restricción.

Figura 01: enlazador
Después de la ligadura, el ADN se restringe nuevamente con enzimas de restricción para producir extremos cohesivos. Ecori-Linkers y los enlazadores SAL-I son enlazadores comúnmente utilizados.
¿Qué es un adaptador??
Un adaptador es una secuencia de oligonucleótidos de doble cadena utilizada para unir dos moléculas de ADN juntas. Es una secuencia corta con un extremo roma y un extremo pegajoso o cohesivo. Por lo tanto, consiste en una cola monocatenaria en un extremo, lo que mejora la eficiencia de la ligadura de ADN.
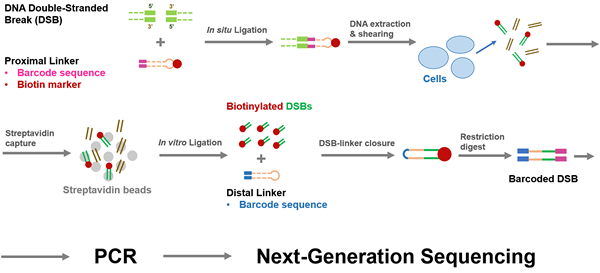
Figura 02: Ligación de ADN por un adaptador
Además, el adaptador tiene sitios de restricción internos. Por lo tanto, después de la ligadura, el ADN puede restringirse con las enzimas de restricción apropiadas para crear un nuevo terminal sobresaliente. Una desventaja de los adaptadores es que dos adaptadores pueden formar atenuadores mediante el emparejamiento de bases con ellos mismos. Esto se puede evitar tratando con la enzima llamada fosfatasa alcalina.
¿Cuáles son las similitudes entre el enlazador y el adaptador??
- Tanto el enlazador como el adaptador son secuencias de oligonucleótidos cortos de doble cadena.
- Llevan sitios de restricción interna.
- Además, son moléculas de ADN sintetizadas químicamente y son moléculas sintéticas.
- Pueden vincular dos moléculas de ADN juntas.
- Después de la ligadura de enlazadores y adaptadores, el ADN se restringe nuevamente con enzimas de restricción para producir extremos pegajosos.
¿Cuál es la diferencia entre enlazador y adaptador??
Un enlazador es un dúplex de oligonucleótido corto sintetizado químicamente con dos extremos contundentes. Un adaptador es un dúplex de oligonucleótido corto sintetizado químicamente con un extremo adhesivo y un extremo roma. Por lo tanto, esta es la diferencia clave entre el enlazador y el adaptador. Además, los adaptadores pueden formar dímeros, mientras que los enlazadores no forman dímeros. Entonces, esta es otra diferencia significativa entre el enlazador y el adaptador.
A continuación se muestra un resumen de las diferencias entre el enlazador y el adaptador en forma tabular.
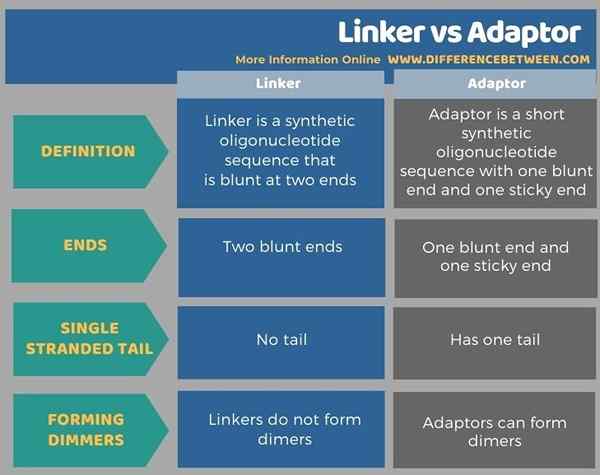
Resumen -Enlazador vs adaptador
El enlazador y el adaptador son dos tipos de oligonucleótidos sintetizados químicamente que son útiles para ligar el ADN de extremo contundente. Linker tiene dos extremos rectos, mientras que el adaptador tiene un extremo roma y un extremo cohesivo. Entonces, esta es la diferencia clave entre el enlazador y el adaptador. Son moléculas de doble cadena que tienen sitios de restricción interna. Se utilizan ampliamente en tecnología de ADN recombinante y clonación de ADN.
Referencia:
1. "ADN de enlazador."Wikipedia, Fundación Wikimedia, 31 de agosto. 2020, disponible aquí.
2. Rothstein, R.J., et al. “[7] Adaptadores sintéticos para clonar ADN."Métodos en enzimología, prensa académica, 7 de enero. 2004, disponible aquí.
Imagen de cortesía:
1. "Bless Workflow (Actualización V2)" de Karahavet - Trabajo propio (CC BY -SA 4.0) a través de Commons Wikimedia
2. "Preparación de la biblioteca para la plataforma sólida" de Philippe Hupé-Emmanuel Barillot, Laurence Calzone, Philippe Hupé, Jean-Philippe Vert, Andrei Zinovyev, Computational Systems Biology of Cancer Chapman & Hall/CRC Biología matemática y computacional, 2012 (CC By-SA-SA 3.0) a través de Commons Wikimedia