Diferencia entre la secuenciación de microarrays y ARN
Diferencia de clave: secuenciación de ARN de microarrays vs
El transcriptoma representa todo el contenido de ARN presente en una célula que incluye ARNm, ARNm, ARNt, ARN degradado y ARN no degradado. El transcriptoma de perfil es un proceso importante para comprender las ideas celulares. Existen varios métodos avanzados para el perfil de transcriptoma. La secuenciación de microarrays y ARN son dos tipos de tecnologías desarrolladas para analizar el transcriptoma. La diferencia clave entre la secuenciación de microarrays y el ARN es que Microarray se basa en el potencial de hibridación de las sondas marcadas prediseñadas con secuencias de ADNc objetivo, mientras que la secuenciación de ARN se basa en la secuenciación directa de las cadenas de ADNc mediante técnicas de secuenciación avanzada como NGS como NGS como NGS. Microarray se realiza con el conocimiento previo sobre las secuencias y la secuenciación de ARN se realiza sin el conocimiento previo sobre las secuencias.
CONTENIDO
1. Descripción general y diferencia de claves
2. Que es microarray
3. ¿Qué es la secuenciación de ARN?
4. Comparación de lado a lado: secuenciación de ARN de microarrays vs
5. Resumen
Que es microarray?
Microarray es un método robusto, confiable y de alto rendimiento utilizado para el perfil de transcriptoma por científicos. Es el enfoque más popular para el análisis de transcripciones. Es un método de bajo costo, que depende de las sondas de hibridación.
La técnica comienza con la extracción de ARNm de la muestra y la construcción de la biblioteca de ADNc a partir de ARN total. Luego se mezcla con sondas prediseñadas marcadas con fluorescencia en una superficie sólida (matriz de manchas). Las secuencias complementarias hibridan con las sondas marcadas en la microarray. Entonces el microarray se lava y se detiene, y la imagen se cuantifica. Los datos recopilados deben analizarse para obtener los perfiles de expresión relativa.
Se supone que la intensidad de las sondas de microarrays es proporcional a la cantidad de transcripciones en la muestra. Sin embargo, la precisión de la técnica depende de las sondas diseñadas, el conocimiento previo de la secuencia y la afinidad de las sondas para la hibridación. Por lo tanto, la tecnología de microarrays tiene limitaciones. La técnica de microarrays no se puede realizar con transcripciones de baja abundancia. No puede diferenciar las isoformas e identificar variantes genéticas. Dado que este método depende de la hibridación de las sondas, algunos problemas relacionados con la hibridación, como la hibridación cruzada, la hibridación inespecífica, etc. ocurre en la técnica de microarrays.

Figura 01: Microarray
¿Qué es la secuenciación de ARN??
Secuenciación de escopeta de ARN (ARN SEQ) es una técnica de secuenciación de transcriptoma completo recientemente desarrollado. Es un método de rendimiento rápido y alto de perfil de transcriptoma. Cuantifica directamente la expresión de genes y da como resultado una investigación profunda del transcriptoma. RNA SEQ no depende de las sondas prediseñadas o el conocimiento previo de las secuencias. Por lo tanto, el método de ARN SEQ tiene una alta sensibilidad y capacidad de detectar genes novedosos y variantes genéticas.
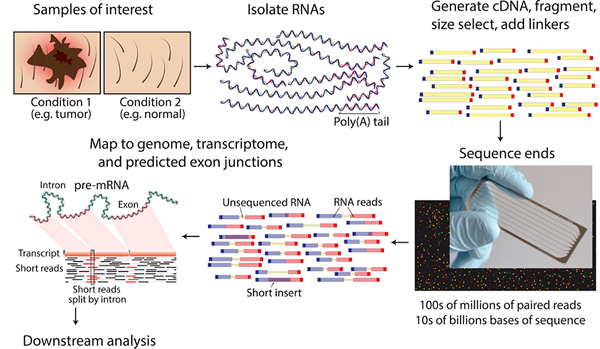
El método de secuenciación de ARN se realiza a través de varios pasos. El ARN total de la célula debe aislarse y fragmentarse. Luego, utilizando la transcriptasa inversa, se debe preparar una biblioteca de ADNc. Cada hilo de ADNc debe ligarse con adaptadores. Entonces los fragmentos ligados deben amplificarse y purificarse. Finalmente, utilizando un método NGS, la secuenciación del ADNc debe realizarse.

Figura 02: secuenciación de ARN
¿Cuál es la diferencia entre la secuenciación de microarrays y ARN??
Secuenciación de microarrays vs ARN | |
| Microarray es un método robusto, confiable y de alto rendimiento. | La secuenciación de ARN es un método preciso y de alto rendimiento. |
| Costo | |
| Este es un método de bajo costo. | Este es un método costoso. |
| Análisis de una gran cantidad de muestras | |
| Esto facilita el análisis de una gran cantidad de muestras simultáneamente. | Esto facilita el análisis de una gran cantidad de muestras. |
| Análisis de los datos | |
| El análisis de datos es complejo. | Se generan más datos en este método; Por lo tanto, el proceso es más complejo. |
| Conocimiento previo de secuencias | |
| Este método se basa en sondas de hibridación, por lo que el conocimiento previo de las secuencias en. | Este método no depende del conocimiento de la secuencia anterior. |
| Variaciones estructurales y genes novedosos | |
| Este método no puede detectar variaciones estructurales y genes novedosos. | Este método puede detectar variaciones estructurales como la fusión de genes, el empalme alternativo y los genes novedosos. |
| Sensibilidad | |
| Esto no puede detectar diferencias en la expresión de isoformas, por lo que esto tiene una sensibilidad limitada. | Esto tiene alta sensibilidad. |
| Resultado | |
| Esto solo puede dar lugar a niveles de expresión relativa. Esto no da cuantificación absoluta de la expresión génica. | Da niveles de expresión absolutos y relativos. |
| Reanálisis de datos | |
| Esto debe volver a la vez para reanalizar. | Los datos de secuenciación se pueden volver a analizar. |
| Necesidad de personal e infraestructura específicos | |
| No se requieren infraestructura y personal específicos para microarrays. | Infraestructura y personal específicos requeridos por la secuenciación de ARN. |
| Problemas técnicos | |
| La técnica de microarrays tiene problemas técnicos como hibridación cruzada, hibridación inespecífica, tasa de detección limitada de sondas individuales, etc. | La técnica de SEQ de ARN evita problemas técnicos como la hibridación cruzada, hibridación inespecífica, tasa de detección limitada de sondas individuales, etc. |
| Sesgo | |
| Este es un método sesgado ya que depende de la hibridación. | El sesgo es bajo en comparación con la microarray. |
Resumen -Secuenciación de ARN de microarray vs
Los métodos de secuenciación de microarrays y ARN son plataformas de alto rendimiento desarrolladas para perfiles de transcriptoma. Ambos métodos producen resultados que están altamente correlacionados con los perfiles de expresión génica. Sin embargo, la secuenciación de ARN tiene ventajas sobre microarrays para el análisis de expresión génica. La secuenciación de ARN es un método más sensible para la detección de transcripciones de baja abundancia que el microarray. La secuenciación de ARN también permite la diferenciación entre las isoformas y la identificación de las variantes genéticas. Sin embargo, Microarray es la elección común de la mayoría de los investigadores, ya que la secuenciación de ARN es una técnica nueva y costosa con desafíos de almacenamiento de datos y análisis de datos complejos.
Referencias:
1.Wang, Zhong, Mark Gerstein y Michael Snyder. "RNA-seq: una herramienta revolucionaria para transcriptómica."Reseñas de la naturaleza. Genética. U.S. Biblioteca Nacional de Medicina, Jan. 2009. Web. 14 mar. 2017
2.Rogler, Charles E., Tatyana Tchaikovskaya, Raquel Norel, Aldo Massimi, Christopher Plescia, Eugeny Rubashevsky, Paul Siebert y Leslie E. Rogler. "Microarrays de expresión de ARN (REMS), un método de alto rendimiento para medir las diferencias en la expresión génica en diversas muestras biológicas."Investigación de ácidos nucleicos. Oxford University Press, 01 de enero. 2004. Web. 15 de mar. 2017
3.Zhao, Shanrong, Wai-Ping Fung-Leung, Anton Bittner, Karen Ngo y Xuejun Liu. "Comparación de ARN-seq y microarrays en el perfil de transcriptoma de las células T activadas." MÁS UNO. Biblioteca Pública de Ciencias, Jan. 2014. Web. 15 de mar. 2017
Imagen de cortesía:
1. "Diario.PCBI.1004393.G002 "por Malachi Griffith, Jason R. Walker, Nicholas C. Espías, Benjamin J. Ainscough, obi l. Griffith - (CC por 2.5) Vía Commons Wikimedia
2. "Microarray" de Bill Branson (fotógrafo) - Instituto Nacional del Cáncer (dominio público) a través de Commons Wikimedia