Diferencia entre reparación de desajuste y reparación de escisión de nucleótidos
Diferencia clave: reparación de desajuste frente a la reparación de escisión de nucleótidos
Decenas y miles de daños de ADN ocurren en la célula por día. Induce cambios en los procesos celulares como la replicación, la transcripción y la viabilidad de la celda. En algunos casos, las mutaciones causadas por estos daños de ADN pueden conducir a enfermedades nocivas como cánceres y síndromes asociados al envejecimiento (ex: progeria). Independientemente de estos daños, la célula inicia un mecanismo de reparación de cascadas altamente organizado llamado respuestas de daño de ADN. Se han identificado varios sistemas de reparación de ADN en el sistema celular; Estos se conocen como reparación de escisión de base (BER), reparación de desajuste (MMR), reparación de escisión de nucleótidos (NER), reparación de ruptura de doble cadena. La reparación de escisión de nucleótidos es un sistema altamente versátil que reconoce lesiones voluminosas de ADN de distorsión de hélice y las elimina. Por otro lado, la reparación de desajuste reemplaza las bases mal incorporadas durante la replicación. La diferencia clave entre la reparación de desajuste y la reparación de la escisión de nucleótidos es que La reparación por escisión de nucleótidos (NER) se usa para eliminar los dímeros de pirimidina formados por la irradiación UV y las lesiones de hélice voluminosas causadas por aductos químicos, mientras que el sistema de reparación de desajuste juega un papel importante en la corrección de bases erróneas que se han escapado de las enzimas de replicación (ADN polimerasa 1) durante la posteplicación durante. Además de las bases no coincidentes, las proteínas del sistema MMR también pueden reparar los bucles de inserciones/ deleciones (IDL) que son resultados del deslizamiento de la polimerasa durante la replicación de secuencias de ADN repetitivas.
CONTENIDO
1. Descripción general y diferencia de claves
2. ¿Qué es la reparación de coincidencias?
3. ¿Qué es la reparación de escisión de nucleótidos?
4. Comparación de lado a lado: reparación de desajuste frente a la reparación de escisión de nucleótidos
5. Resumen
¿Qué es la reparación de escisión de nucleótidos??
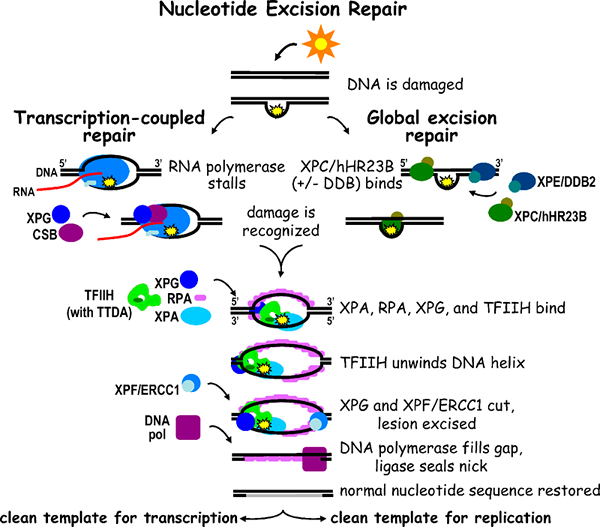
La característica más distinguida de la reparación de la escisión de nucleótidos es que repara los daños de nucleótidos modificados causados por distorsiones significativas en la doble hélice de ADN. Se observa en casi todos los organismos que se han examinado hasta la fecha. UVR A, UVR B, UVR C (excinucleasas) UVR D (una helicasa) son las enzimas más conocidas involucradas en el NER que desencadenan la reparación de ADN en el organismo modelo Ecoli. El complejo enzimático multisuperiño UVR ABC produce los polipéptidos UVR A, UVR B, UVR C. Los genes codificados para los polipéptidos mencionados son UVR A, UVR B, UVR C. Las enzimas UVR A y B reconocen colectivamente la distorsión inducida por el daño que se causa a la doble hélice de ADN, como los atenuadores de pirimidina debido a la irradiación UV. UVR A es una enzima ATPasa y esta es una reacción autocatalítica. Luego, la UVR A deja el ADN, mientras que el complejo UVR BC (nucleasa activa) escinde el ADN en ambos lados del daño que se cataliza por ATP. Otra proteína llamada UVR D codificada por el gen UVRD es una enzima Helicasa II relájate el ADN que resulta de la liberación del segmento de ADN dañado por un solo cadáver. Esto deja un espacio en la hélice de ADN. Después de que se ha extirpado el segmento dañado, queda un espacio de nucleótidos de 12-13 en la cadena de ADN. Esto se llena por la enzima ADN polimerasa I y la muesca está sellada por la ADN ligasa. Se requiere ATP en tres pasos de esta reacción. El mecanismo ner también se puede identificar en los humanos como los mamíferos. En los humanos, la condición de la piel llamada Xeroderma pigmentosum se debe a los dímeros de ADN causados por la irradiación UV. Los genes XPA, XPB, XPC, XPD, XPE, XPF y XPG producen proteínas para reemplazar el daño del ADN. Las proteínas de los genes XPA, XPC, XPE, XPF y XPG tienen la actividad de nucleasa. Por otro lado, las proteínas de los genes XPB y XPD muestran la actividad de la helicasa que anátela a UVR D en E. coli.

Figura 01: Reparación de escisión de nucleótidos
¿Qué es la reparación de coincidencias??
El sistema de reparación de desajuste se inicia durante la síntesis de ADN. Incluso con la subunidad € funcional, la ADN polimerasa III permite la incorporación de un nucleótido incorrecto para la síntesis cada 108 pares de bases. Las proteínas de reparación de desajuste reconocen este nucleótido, lo improduzcan y reemplácelo con el nucleótido correcto responsable del grado final de precisión. La metilación del ADN es fundamental para que las proteínas MMR reconozcan el hilo principal de la cadena recientemente sintetizada. La metilación del nucleótido adenina (a) en un motivo GATC de un hilo recientemente sintetizado se retrasa un poco. Por otro lado, el nucleótido de adenina de la hebra padre en el motivo GATC ya ha metilado. Las proteínas MMR reconocen el hilo recientemente sintetizado por esta diferencia con el hilo principal y comienzan la reparación de desajuste en un hilo recientemente sintetizado antes de que se metilice. Las proteínas MMR dirigen su actividad de reparación para eliminar el nucleótido incorrecto antes de que la cadena de ADN recién replicado se metilice. Las enzimas Mut H, Mut L y Mut s codificadas por genes Mut H, Mut L, Mut S catalizan estas reacciones en Ecoli. La proteína Mut S reconoce siete de ocho pares de bases de desajuste posibles, excepto C: C, y se une en el sitio de desajuste en el ADN dúplex. Con ATP unidos, Mut L y Mut s se unen al complejo más tarde. El complejo transloca a pocos miles de pares de bases hasta que encuentre un motivo GATC hemimetilado. La actividad inactiva de la nucleasa de la proteína Mut H se activa una vez que encuentra un motivo GATC hemimetilado. Cinta la cadena de ADN no metilada dejando una nick 5 'en el nucleótido G del motivo GATC no metilado (cadena de ADN recién sintetizada). Entonces el mismo hilo en el otro lado del desajuste es mordido por Mut H. En el resto de los pasos, las acciones colectivas de UVR d a proteína helicasa, Mut U, SSB y exonucleasa I implican el nucleótido incorrecto en el ADN monocatenario. La brecha que se forma en la escisión se llena por la ADN polimerasa III y sellada por ligasa. Se puede identificar un sistema similar en ratones y humanos. La mutación de HMLH1, HMSH1 y HMSH2 humanos está involucrada en el cáncer de colon no poliposis hereditaria que desregula la división celular de las células de colon.
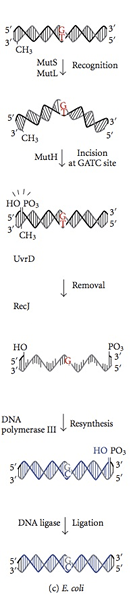
Figura 02: reparación de desajuste
¿Cuál es la diferencia entre la reparación de desajuste y la reparación de escisión de nucleótidos??
Reparación de desajuste vs reparación de escisión de nucleótidos | |
| El sistema de reparación de desajuste ocurre durante la replicación posterior. | Esto está involucrado en la eliminación de dímeros de pirimidina debido a u.V Irradiación y otras lesiones de ADN debido al aducto químico. |
| Enzimas | |
| Es catalizado por Mut S, Mut L, Mut H, UVR D, SSB y Exonuclease I. | Es catalizado por UVR A, UVR B, UVR C, enzimas UVRD. |
| Metilación | |
| Es fundamental iniciar la reacción. | La metilación del ADN no es necesaria para iniciar la reacción. |
| Acción de las enzimas | |
| Mut H es una endonucleasa. | UVR B y UVR C son exonucleasas. |
| Ocasión | |
| Esto sucede específicamente durante la replicación. | Esto sucede cuando se expone a ti.V o mutágenos químicos, no durante la replicación |
| Conservación | |
| Está altamente conservado | No está muy conservado. |
| Llenado de brechas | |
| Se realiza por ADN polimerasa III. | Se hace por la ADN polimerasa I. |
Resumen: reparación de desajuste frente a la reparación de escisión de nucleótidos
La reparación de desajuste (MMR) y la reparación de escisión de nucleótidos (NER) son dos mecanismos que tienen lugar en la célula para rectificar los daños y distorsiones de ADN causados por varios agentes. Estos se denominan colectivamente como mecanismos de reparación de ADN. Reparaciones de reparación de escisión de nucleótidos Los daños modificados de nucleótidos, típicamente esos daños significativos de la doble hélice de ADN que ocurren debido a la exposición a U.V Irradiación y aductos químicos. Las proteínas de reparación de desajuste reconocen el nucleótido incorrecto, lo impulsan y lo reemplazan con nucleótido correcto. Este proceso es responsable del grado final de precisión durante la replicación.
Referencia:
1.Cooper, Geoffrey M. "Reparación de ADN."La célula: un enfoque molecular. 2ª edición.U.S. Biblioteca Nacional de Medicina, 01 de enero. 1970. Web. 09 mar. 2017.
2."Mecanismos y funciones de la reparación del desajuste de ADN."Investigación celular. U.S. Biblioteca Nacional de Medicina, N.d. Web. 09 mar. 2017.
Imagen de cortesía:
1. "Reparación de escisión de nucleótidos-Journal.pbio.0040203.G001 ”por Jill O. Alboroto, Priscilla K. Cooper - (CC por 2.5) Vía Commons Wikimedia
2. "Ecoli de reparación de desajuste de ADN" por Kenji Fukui - (CC por 4.0) a través de Commons Wikimedia


