Diferencia entre la secuenciación de NGS y Sanger
Diferencia de clave: secuenciación NGS vs Sanger
La secuenciación de próxima generación (NGS) y la secuenciación de Sanger son dos tipos de técnicas de secuenciación de nucleótidos desarrolladas a lo largo del tiempo. El método de secuenciación de Sanger se usó ampliamente durante muchos años y NGS lo reemplazó recientemente debido a sus ventajas. La diferencia clave entre la secuencia NGS y Sanger es que El NGS funciona sobre el principio de secuenciación de millones de secuencias simultáneamente de manera rápida a través de un sistema de secuenciación, mientras que la secuenciación de Sanger funciona en el principal de la terminación de la cadena debido a la incorporación selectiva de dideoxinucleótidos por enzima de ADN polimerasa durante la replicación de ADN y la separación de la fragmentación de los resultados mediante capilares capilitarias electroforesis.
CONTENIDO
1. Descripción general y diferencia de claves
2. ¿Qué es la secuenciación de nucleótidos?
3. Que es NGS
4. ¿Qué es la secuenciación de Sanger?
5. Comparación de lado a lado - NGS vs Sanger Sequencing
6. Resumen
¿Qué es la secuenciación de nucleótidos??
La información genética se almacena en las secuencias de nucleótidos del ADN o ARN de un organismo. El proceso de determinar el orden correcto de los nucleótidos (usando cuatro bases) en un fragmento dado (en un gen, el grupo de genes, el cromosoma y el genoma completo) se conoce como secuenciación de nucleótidos. Es muy importante en estudios genómicos, estudios forenses, virología, sistemático biológico, diagnóstico médico, biotecnología y en muchos otros campos para analizar la estructura y la función de los genes. Existen diferentes tipos de métodos de secuenciación desarrollados por científicos. Entre ellos, Secuenciación de Sanger desarrollado por Frederick Sanger en 1977 fue ampliamente utilizado y popularizado durante un largo período hasta Secuenciación de la próxima generación lo reemplazó.
Que es NGS?
La secuenciación de la próxima generación (NGS) es un término utilizado para referirse a los procesos modernos de secuenciación de alto rendimiento. Describe una serie de diferentes tecnologías de secuenciación modernas que revolucionaron los estudios genómicos y la biología molecular. Esas técnicas son la secuenciación de Illumina, la secuenciación de Roche 454, la secuenciación de protones iónicos y la secuenciación Solid (secuenciación por detección de ligadura de oligo). Los sistemas NGS son más rápidos y más baratos. Se utilizan cuatro métodos principales de secuenciación de ADN en sistemas NGS, a saber; Pyrosequencing, secuenciación por síntesis, secuenciación por ligadura y secuenciación de semiconductores de iones. Se puede secuenciar una gran cantidad de hilos de ADN o ARN (millones de). Permite la secuenciación de todo el genoma de los organismos en un corto período de tiempo, a diferencia de la secuenciación de Sanger que lleva más tiempo.
NGS tiene muchas ventajas sobre el método de secuenciación convencional Sanger. Es un proceso de alta velocidad, más preciso y rentable que se puede realizar con un pequeño tamaño de muestra. Los NG se pueden usar en estudios metagenómicos, en la detección de variaciones dentro de un genoma individual debido a inserciones y deleciones, etc. y en el análisis de las expresiones génicas.
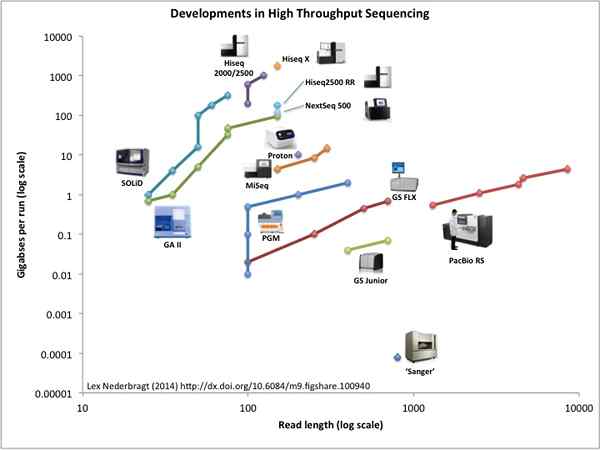
Figura_1: Desarrollos en la secuenciación de NGS
¿Qué es la secuenciación de Sanger??
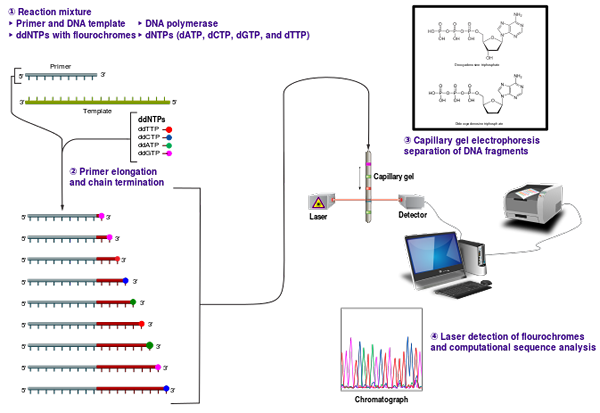
La secuenciación de Sanger es un método de secuenciación desarrollado por Frederick Sanger y sus colegas en 1977 para determinar el orden de nucleótidos preciso de un fragmento de ADN dado. También se conoce como secuenciación de terminación de cadena o Secuenciación de dedoxi. El principio de funcionamiento de este método es la terminación de la síntesis de la cadena mediante la incorporación selectiva de una cadena que termina los dedoxinucleótidos (DDNTP) como DDGTP, DDCTP, DDATP y DDTTP por ADN polimerasa durante la replicación de ADN. Los nucleótidos normales tienen 3 grupos 'OH para la formación de un enlace fosfodiéster entre los nucleótidos adyacentes para continuar la formación de la cadena. Sin embargo, los DDNTP carecen de este grupo 3 'OH y no pueden formar enlaces fosfodiesteros entre los nucleótidos. Por lo tanto, el alargamiento de la cadena ha cesado.
En este método, el ADN monocatenario a secuenciar sirve como la cadena de plantilla para in vitro Síntesis de ADN. Otros requisitos son el cebador de oligonucleótidos, los precursores de desoxinucleótidos y la enzima de ADN polimerasa. Cuando se conocen los extremos flanqueantes del fragmento objetivo, los cebadores se pueden diseñar fácilmente para la replicación del ADN. Se realizan cuatro reacciones de síntesis de ADN separadas en cuatro tubos separados. Cada tubo tiene ddntps separados, junto con otros requisitos. Del nucleótido particular, se agregan una mezcla de dntps y ddntps. Del mismo modo, se realizan cuatro reacciones separadas en cuatro tubos con cuatro mezclas. Después de las reacciones, se realizan la detección de fragmentos de ADN y la conversión del patrón de fragmento en información de secuencia. Los fragmentos de ADN resultantes son desnaturalizados por calor y se separan por electroforesis en gel. Si se usan nucleótidos radiactivos, el patrón de bandas en el gel de poliacrilamida se puede visualizar mediante autorradiografía. Cuando este método utiliza los dedoxinucleótidos etiquetados con fluorescencia, se puede mitigar el gel leído y pasar a través de un haz de láser para ser detectado por el detector fluorescente. Para evitar errores que puedan surgir cuando el ojo lea una secuencia e ingrese manualmente en una computadora, este método se convirtió en el uso de secuenciador automatizado junto con la computadora.
Este es el método utilizado para secuenciar el ADN del proyecto del genoma humano. Este método todavía está en uso con modificaciones avanzadas porque proporciona información de secuencia precisa a pesar de ser un proceso costoso y lento.

Figura_2: secuenciación de Sanger
¿Cuál es la diferencia entre la secuenciación de NGS y Sanger??
Secuenciación NGS vs Sanger | |
| La secuenciación de la próxima generación (NGS) consulte los procesos modernos de secuenciación de alto rendimiento. Describe una serie de tecnologías de secuenciación modernas diferentes | La secuenciación de Sanger es un método de secuenciación desarrollado por Frederick Sanger para determinar el orden de nucleótidos preciso de un fragmento de ADN dado. |
| Rentabilidad | |
| NGS es un proceso más barato porque reduce el tiempo, la potencia del hombre y los productos químicos. | Este es un proceso costoso porque lleva tiempo, potencia del hombre y más productos químicos. |
| Velocidad | |
| Esto es más rápido ya que tanto la detección química como la detección de señales de muchos hilos están ocurriendo paralelo. | Esto lleva mucho tiempo ya que la detección de productos químicos y la detección de señales ocurren como dos procesos separados y solo en la cadena pueden leer a la vez. |
| Fiabilidad | |
| NGS es confiable. | La secuenciación de Sanger es menos confiable |
| Tamaño de la muestra | |
| NGS requiere menos cantidad de ADN. | Este método necesita una gran cantidad de ADN de plantilla. |
| Bases de ADN por fragmento secuenciado | |
| El número de bases de ADN por fragmento secuenciado es menor que el método Sanger | Las secuencias generadoras son más largas que las secuencias NGS. |
Resumen - Ngs vs Sanger Secuenciación
La secuenciación NGS y Sanger son técnicas de secuenciación de nucleótidos ampliamente utilizadas en biología molecular. La secuenciación de Sanger es un método de secuenciación temprana que fue reemplazado por NGS. La principal diferencia entre la secuenciación de NGS y Sanger es que NGS es un proceso de alta velocidad, más preciso y rentable que la secuenciación de Sanger. Ambas técnicas crearon brotes importantes en genética y biotecnología.
Referencia:
1. Nowrousian, Minou. "Técnicas de secuenciación de próxima generación para microorganismos eucariotas: soluciones basadas en secuenciación a problemas biológicos." Célula eucariota. Sociedad Americana de Microbiología, septiembre. 2010. Web. 18 de febrero. 2017
2. Sanger, F., S. Nicklen y un. Riñonal. Coulson. “Securificación de ADN con inhibidores de terminación de cadena.Actas de la Academia Nacional de Ciencias 74.12 (1977): 5463-467. Web.
3. Liu, Lin, Yinhu Li, Siliang Li, Ni Hu, Yimin He, Ray Pong, Danni Lin, Lihua Lu y Maggie Law. "Comparación de sistemas de secuenciación de próxima generación."Journal of Biomedicine and Biotechnology 2012 (2012): 1-11. Web.
Imagen de cortesía:
"Sanger-Securencing" de Estevezj-Trabajo propio (CC By-SA 3.0) a través de Commons Wikimedia
"Desarrollos en la secuenciación de la próxima generación" por Nederbragt, Lex (2012) - (CC por 3.0) a través de Commons Wikimedia