Diferencia entre la secuenciación de Sanger y la pirosequenciación
Diferencia de clave: secuenciación de Sanger vs Pyrosequencing
La secuenciación de ADN es muy importante para el análisis de ADN, ya que el conocimiento de la disposición de los nucleótidos correctos en una región de ADN particular revela mucha información importante al respecto. Hay diferentes métodos de secuenciación de ADN. La secuenciación y la pirosia de Sanger son dos métodos de secuenciación de ADN diferentes ampliamente utilizados en la biología molecular. La diferencia clave entre la secuenciación de Sanger y la pirosequenciación es que La secuenciación de Sanger utiliza dedoxinucleótidos para terminar la síntesis de ADN para leer la secuencia de nucleótidos mientras la pirosequenciación detecta la liberación de pirofosfato al incorporar los nucleótidos y sintetizar la secuencia complementaria para leer el orden preciso de la secuencia.
CONTENIDO
1. Descripción general y diferencia de claves
2. ¿Qué es la secuenciación de Sanger?
3. ¿Qué es la pirosequencing?
4. Comparación de lado a lado: secuenciación de Sanger vs Pyrosequencing
5. Resumen
¿Qué es la secuenciación de Sanger??
La secuencia de Sanger es un método de secuenciación de ADN de primera generación desarrollado por Frederick Sanger y sus universidades en 1977. También se conoce como Secuenciación de terminación de cadena o Secuenciación de dedoxi Dado que se basa en la terminación de la cadena por dideoxinucleótidos (DDNTP). Este método se usó ampliamente durante más de 30 años hasta que se desarrolló la secuenciación de la nueva generación (NGS). La técnica de secuenciación de Sanger permitió el descubrimiento del orden de nucleótidos correcto o la unión de un fragmento de ADN particular. Se basa en la incorporación selectiva de DDNTP y la terminación de la síntesis de ADN durante el in vitro Replicación de ADN. La ausencia de 3 grupos 'OH para continuar la formación de enlaces de fosfodiéster entre nucleótidos adyacentes es una característica única de DDNTPS. Por lo tanto, una vez que se une el DDNTP, el alargamiento de la cadena cesa y termina desde ese punto. Hay cuatro DDNTP - DDATP, DDCTP, DDGTP y DDTTP - utilizados en la secuenciación de Sanger. Estos nucleótidos detienen el proceso de replicación del ADN cuando se incorporan a la hebra creciente de ADN y dan como resultado longitudes variables de ADN corto. La electroforesis en gel capilar se usa para organizar estos hilos de ADN cortos por sus tamaños en un gel como se muestra en la Figura 01.
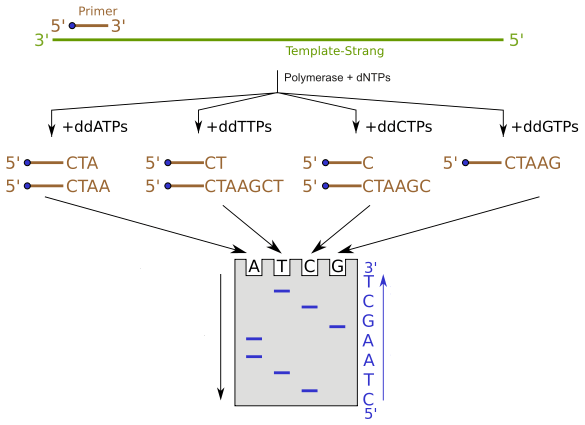
Figura 1: electroforesis en gel capilar de ADN corto sintetizado
Para in vitro Replicación de ADN, se deben proporcionar pocos requisitos. Son enzima ADN polimerasa, ADN de plantilla, cebadores de oligonucleótidos y desoxinucleótidos (DNTP). En la secuenciación de Sanger, la replicación de ADN se realiza en cuatro tubos de ensayo separados junto con cuatro tipos de DDNTP por separado. Los desoxinucleótidos no son totalmente reemplazados por los respectivos ddntps. Una mezcla del DNTP particular (por ejemplo; DATP + DDATP) se incluye en el tubo y se replica. Cuatro productos de tubo separados se ejecutan en un gel en cuatro pozos separados. Luego, al leer el gel, la secuencia se puede construir como se muestra en la Figura 02.

Figura 02: secuenciación de Sanger
La secuenciación de Sanger es una técnica importante que ayuda en muchas áreas de biología molecular. El proyecto del genoma humano se completó con éxito con la ayuda de métodos basados en secuenciación de Sanger. La secuenciación de Sanger también es útil en la secuenciación del ADN objetivo, la investigación del cáncer y la enfermedad genética, el análisis de expresión génica, la identificación humana, la detección de patógenos, la secuenciación microbiana, etc.
Hay varias desventajas de la secuenciación de Sanger:
- La longitud del ADN que se secuencia no puede ser mayor que 1000 pares de bases
- Solo se puede secuenciar un hilo a la vez.
- El proceso lleva mucho tiempo y costoso.
Por lo tanto, se desarrollaron nuevas técnicas de secuenciación avanzada con tiempo para superar estos problemas. Sin embargo, la secuenciación de Sanger todavía está en uso debido a sus resultados altamente precisos hasta aproximadamente 850 fragmentos de pareja de bases.
¿Qué es la pirosequencing??
Pyrosequencing es una nueva técnica de secuenciación de ADN basada en la "secuenciación por síntesis". Esta técnica se basa en la detección de la liberación de pirofosfato en la incorporación de nucleótidos. El proceso es empleado por cuatro enzimas diferentes: ADN polymerse, sulfurilasa de ATP, luciferasa y apirasa y dos sustratos de fosfosulfato de adenosina 5 '(APS) y luciferina.
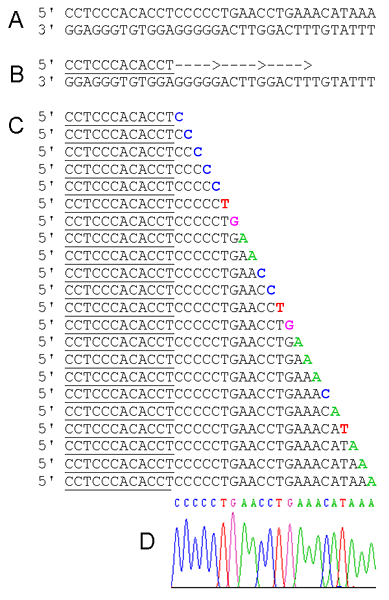
El proceso comienza con la unión del cebador con la plantilla de ADN monocatenaria y la ADN polimerasa inicia la incorporación de nucleótidos complementarios a él. Cuando los nucleótidos se unen (polimerización del ácido nucleico), libera grupos de pirofosfato (dos grupos fosfatos unidos) y energía. Cada adición de nucleótidos libera cantidad equimolar de pirofosfato. El pirofosfato se convierte en ATP por ATP sulfurilasa en presencia de APS sustrato. El ATP generado impulsa la conversión mediada por luciferasa de luciferina a oxiluciferina, produciendo luz visible en cantidades que son proporcionales a la cantidad de ATPS. La luz es detectada por un dispositivo de detección de fotones o por fotomultiplicador y crea un pirograma. La apirasa degrada ATP y los DNTP no incorporados en la mezcla de reacción. La adición de DNTP se realiza una vez a la vez. Dado que la adición de nucleótido se conoce de acuerdo con la incorporación y detección de la luz, se puede determinar la secuencia de la plantilla. El pirograma se usa para generar la secuencia de nucleótidos del ADN de la muestra como se muestra en la Figura 03.
La pirosequenciación es muy importante en el análisis de polimorfismo de un solo nucleótido y la secuenciación de tramos cortos de ADN. La alta precisión, la flexibilidad, la facilidad de automatización y el procesamiento paralelo son las ventajas de la pirosequenciación sobre las técnicas de secuenciación de Sanger.
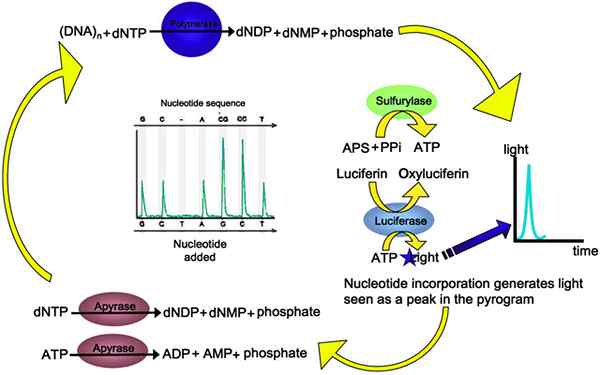
Figura 03: Pyrosequencing
¿Cuál es la diferencia entre la secuenciación de Sanger y la pirosequenciación??
Secuenciación de sanger vs pirosequencing | |
| La secuenciación de Sanger es un método de secuenciación de ADN basado en la incorporación selectiva de DDNTP por la ADN polimerasa y la terminación de la cadena. | Pyrosequencing es un método de secuenciación de ADN basado en la detección de la liberación de pirofosfato tras la incorporación de nucleótidos. |
| Uso de ddntp | |
| Los DDNTP se utilizan para terminar la replicación del ADN | Los ddntps no se usan. |
| Enzimas involucradas | |
| Se usan la ADN polimerasa. | Se utilizan cuatro enzimas: ADN polimerasa, ATP sulfurilasa, luciferasa y apirasa. |
| Sustratos utilizados | |
| APS y Luciferin no se usan. | Se usan fosfosulfato de adenosina 5 '(APS) y luciferina. |
| Temperatura máxima | |
| Este es un proceso lento. | Este es un proceso rápido. |
Resumen: secuenciación de Sanger vs Pyrosequencing
La secuenciación y la pirosequenciación de Sanger son dos métodos de secuenciación de ADN utilizados en la biología molecular. La secuenciación de Sanger construye el orden de los nucleótidos en la secuencia terminando el alargamiento de la cadena, mientras que la pirosequenciación construye el orden preciso de los nucleótidos en la secuencia mediante la incorporación de nucleótidos y detectando la liberación de pirofosfatos. Por lo tanto, la principal diferencia entre la secuenciación de Sanger y la pirosequenciación es que la secuenciación de Sanger funciona en la secuenciación por terminación de la cadena, mientras que la pirosequenciación funciona en la secuenciación por síntesis.
Referencia:
1. Fakruddin, MD y Abhijit Chowdhury. "Pyrosequencing: una alternativa a la secuencia tradicional de Sanger."American Journal of Biochemistry and Biotechnology. Publicaciones de ciencias, 02 mar. 2012. Web. 28 de febrero. 2017.
2. "Sugencia de Sanger."Sectrencing Sanger - Temas de ciencias de las ciencias. norte.pag., norte.d. Web. 28 de febrero. 2017
Imagen de cortesía:
1. "Didesoxi -Methode" de Christoph Goemans (modifiziert) - Dr. Norman Mauder, Auf Base Einer Datei von Christoph Goemans (CC By-SA 3.0) a través de Commons Wikimedia
2. "Sanger-Dna-seq" de Enzo en la lengua polaca Wikipedia (CC By-SA 3.0) a través de Commons Wikimedia
3. "Pyrosequencing" de MicrobiologyBytes (CC BY-SA 2.0) a través de Flickr