Diferencia entre la secuenciación de la escopeta y la secuenciación de la próxima generación
El diferencia clave Entre la secuenciación de escopeta y la secuenciación de la próxima generación es que La secuenciación de la escopeta es un método de secuenciación que rompe aleatoriamente las secuencias de ADN en muchos pequeños fragmentos y vuelve a ensamblar la secuencia al observar las regiones superpuestas, mientras que la próxima secuenciación de la próxima generación (NGS) es un método avanzado de secuenciación genética que depende de la electroforesis capilar.
La secuenciación es el proceso que determina el orden preciso de los nucleótidos en un gen, un grupo de genes, cromosomas y un genoma completo. Es muy importante en estudios genómicos, estudios forenses, virología, sistemático biológico, diagnóstico médico, biotecnología y en muchos otros campos para analizar la estructura y la función de los genes e identificación de organismos. Además, existen diferentes tipos de métodos de secuenciación disponibles. La secuenciación de escopeta y la secuenciación de la próxima generación son dos métodos avanzados entre ellos.
CONTENIDO
1. Descripción general y diferencia de claves
2. ¿Qué es la secuencia de escopeta?
3. ¿Qué es la secuenciación de la próxima generación?
4. Similitudes entre la secuenciación de la escopeta y la secuenciación de la próxima generación
5. Comparación lado a lado: secuenciación de escopeta frente a la secuenciación de la próxima generación en forma tabular
6. Resumen
¿Qué es la secuencia de escopeta??
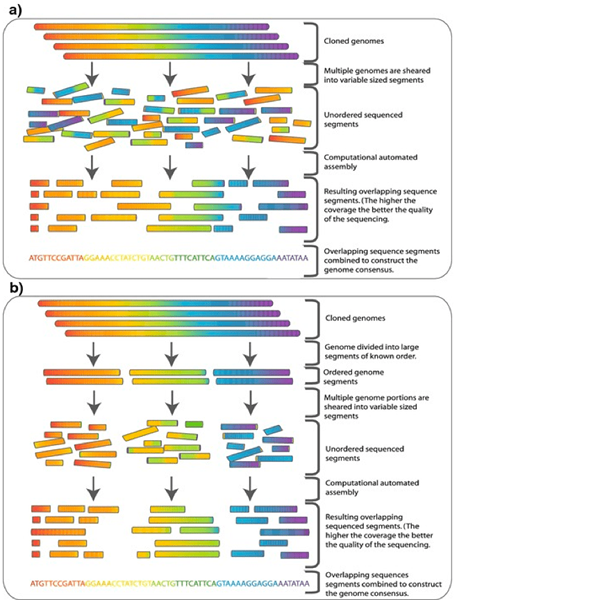
La secuenciación de escopeta es un método de secuenciación que rompe aleatoriamente las secuencias de ADN de todo el cromosoma o genoma completo en muchos fragmentos pequeños y vuelve a montar las secuencias por las computadoras observando las secuencias o regiones superpuestas. En general, los genomas de los mamíferos son estructuralmente complejos y de mayor tamaño. Por lo tanto, son difíciles de secuencia por clonación ya que lleva mucho tiempo. La secuenciación de escopeta es un método más rápido. También es más barato llevar a cabo. Por lo tanto, los científicos modernos confían en el método de secuenciación de escopetas para abordar genomas complejos.

Figura 01: secuenciación de escopeta
El procedimiento de secuenciación de escopeta es relativamente simple. Comienza fragmentando todo el genoma en diferentes tamaños de bases de 20 kilo a bases de 300 kilo. Entonces todos y cada uno de los fragmentos deben secuenciarse utilizando el método de terminación de la cadena. Después de la secuenciación, es necesario ensamblar los fragmentos mirando las regiones superpuestas utilizando sofisticado software de computadora. El mapeo convencional y la clonación de secuencias no son necesarios para este método. Además, el uso del mapa genético no tiene lugar en este método. Sin embargo, dado que no hay uso de los mapas del genoma existentes, es más probable que ocurran errores durante el ensamblaje. Es una de las principales desventajas de este método. Además, los fragmentos más cortos proporcionan información menos única para cada lectura en este método. Además, la secuenciación de escopeta no produce suficientes datos para determinar una secuencia de consenso en el estándar de precisión requerido.
A pesar de tener los inconvenientes mencionados anteriormente, el método de secuenciación de escopeta es actualmente la estrategia más eficiente y rentable para secuenciar genomas microbianos, incluidas bacterias, virus y levadura. Es porque sus genomas carecen de regiones repetitivas que son difíciles de secuencia, y es posible ensamblar estos genomas fácilmente en cromosomas sin errores.
¿Qué es la secuenciación de la próxima generación??
La secuenciación de la próxima generación (NGS) es un término utilizado para referirse a los procesos modernos de secuenciación de alto rendimiento. Describe una serie de diferentes tecnologías de secuenciación modernas que revolucionaron los estudios genómicos y la biología molecular. Estas técnicas incluyen secuenciación de Illumina, secuenciación de Roche 454, secuenciación de protones iónicos y secuenciación sólida (secuenciación por detección de ligadura de oligo). Los sistemas NGS son más rápidos y más baratos. Cuatro métodos principales de secuenciación de ADN se utilizan en los sistemas NGS: pirosequenciación, secuenciación por síntesis, secuenciación por ligadura y secuenciación de semiconductores de iones. Una gran cantidad de hilos de ADN o ARN (millones de) pueden ser secuenciados en paralelo por NGS. Permite la secuenciación de todo el genoma de los organismos en un corto período de tiempo.

Figura 02: secuenciación de la próxima generación
NGS tiene diferentes ventajas. Es un proceso de alta velocidad, más preciso y rentable que se puede realizar con un pequeño tamaño de muestra. Por lo tanto, permite el análisis de todo el genoma humano en un solo experimento de secuenciación. Además, los NG se pueden usar en estudios metagenómicos, en la detección de variaciones dentro de un genoma individual debido a inserciones y deleciones, etc., y en el análisis de las expresiones génicas. Además, NGS puede analizar transcriptomas completos de grandes cantidades de tejidos simultáneamente. Por lo tanto, NGS ha revolucionado el análisis de los transcriptomas.
¿Cuáles son las similitudes entre la secuenciación de la escopeta y la secuenciación de la próxima generación??
- La secuenciación de escopeta y la secuenciación de la próxima generación son dos métodos de secuenciación del genoma.
- Ambos métodos son métodos rápidos.
- Además, son métodos rentables.
- Pueden secuenciar muchos fragmentos de ADN en paralelo.
¿Cuál es la diferencia entre la secuenciación de la escopeta y la secuenciación de la próxima generación??
La secuenciación de escopeta y la secuenciación de la próxima generación son dos técnicas de secuenciación avanzada. El método de secuenciación de escopeta rompe aleatoriamente las secuencias de ADN de todo el cromosoma o todo el genoma en muchos fragmentos pequeños y vuelve a ensamblar las secuencias por las computadoras al observar las secuencias o regiones superpuestas. En contraste, la secuenciación de la próxima generación (NGS) es un término que se refiere a los procesos modernos de secuenciación de alto rendimiento. Entonces, esta es la diferencia clave entre la secuenciación de la escopeta y la secuenciación de la próxima generación. Además, la secuenciación de la próxima generación es una técnica que depende de la electroforesis capilar, mientras que la secuenciación de escopeta no depende. Por lo tanto, esta también es una diferencia entre la secuenciación de la escopeta y la secuenciación de la próxima generación. Además, en comparación con la secuenciación de la escopeta, la secuenciación de próxima generación es altamente sensible y altamente precisa .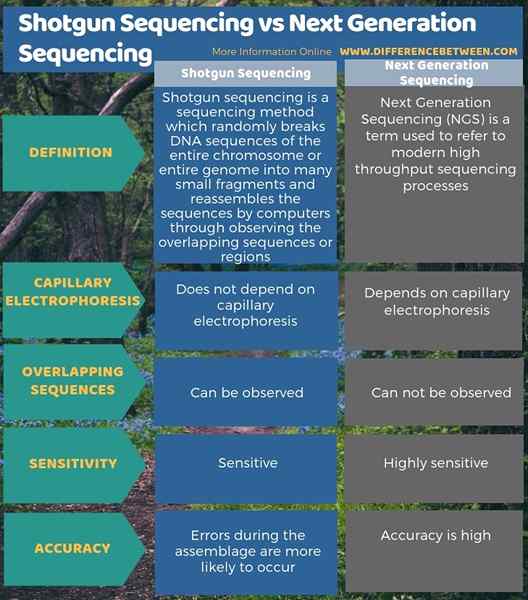
Resumen: secuenciación de escopeta frente a la secuenciación de la próxima generación
La secuenciación de escopeta y la secuenciación de la próxima generación son dos métodos de secuenciación utilizados en la secuenciación del genoma. Ambos métodos son métodos rápidos y rentables. NGS funciona sobre el principio de secuenciar millones de secuencias simultáneamente de manera rápida a través de un sistema de secuenciación. En contraste, la secuenciación de escopeta requiere la ruptura de genomas en pequeños fragmentos y secuenciación y reensambla utilizando secuencias superpuestas. Entonces, esto resume la diferencia entre la secuenciación de la escopeta y la secuenciación de la próxima generación.
Referencia:
1. "Genomas complejos: secuenciación de escopeta" Nature News, Nature Publishing Group, disponible aquí.
2. Behjati, Sam y Patrick S Tarpey. "¿Qué es la secuenciación de la próxima generación??"Archivos de la enfermedad en la infancia. Edición de educación y práctica, BMJ Publishing Group, DEC. 2013, disponible aquí.
Imagen de cortesía:
1. "Secuenciación de escopeta de genoma completo versus secuenciación de escopeta jerárquica" de Commins, J., Toft, c., Tarifas, m. A. - “Métodos de biología computacional y su aplicación a la genómica comparativa de las bacterias simbióticas endocelulares de los insectos."Biol. Procedimientos en línea (2009). Accedido a través de SpringerImages (CC BY-SA 2.5) Vía Commons Wikimedia
2. "Hiseq 2000" por RE73 - Trabajo propio (CC By -SA 3.0) a través de Commons Wikimedia