¿Cuál es la diferencia entre CGH y Array CGH?
El diferencia clave entre CGH y Array CGH es que CGH es un método citogenético molecular tradicional para analizar la variación del número de copias en el ADN de una muestra de prueba que tiene una resolución más baja, mientras que la matriz CGH es un método citogenético molecular totalmente automatizado para analizar la variación del número de copias en el ADN de una muestra de prueba que tiene un resolución más alta.
CGH y Array CGH son dos técnicas para detectar variaciones de número de copias en individuos. La variación del número de copias se refiere a las secciones del genoma que se repiten. El número de copias varía entre individuos. La variación del número de copias generalmente se clasifica en dos grupos: repeticiones cortas y repeticiones largas. Uno de los ejemplos más conocidos para una variación de número de copia corta es la repetición del trinucleótido CAG en el gen huntingtin. Esto causa un trastorno neurológico llamado Huntingtin. El mejor ejemplo para las repeticiones largas de la variación del número de copias es el gen alfa amilasa, que causa resistencia a la insulina. La variación del número de copias puede detectarse mediante técnicas como Fish, CGH y Array CGH.
CONTENIDO
1. Descripción general y diferencia de claves
2. Que es CGH
3. ¿Qué es Array CGH?
4. Similitudes - CGH y Array CGH
5. CGH vs Array CGH en forma tabular
6. Resumen - CGH vs Array CGH
Que es CGH?
CGH es un método citogenético molecular tradicional para analizar la variación del número de copias en el ADN de una muestra de prueba en comparación con una muestra de referencia. Tiene una resolución más baja en comparación con las técnicas automatizadas. El objetivo de esta técnica es comparar de manera rápida y eficiente dos muestras de ADN genómico que surgen de diferentes fuentes.
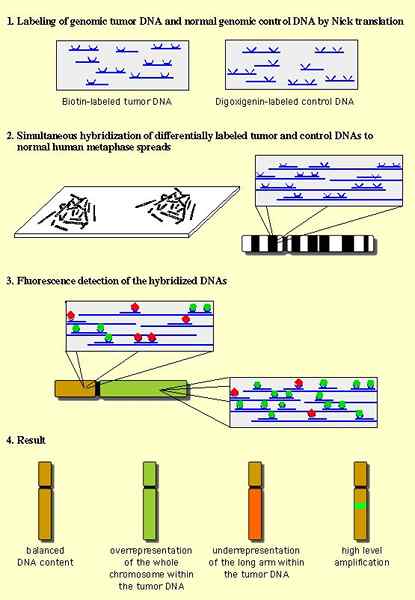
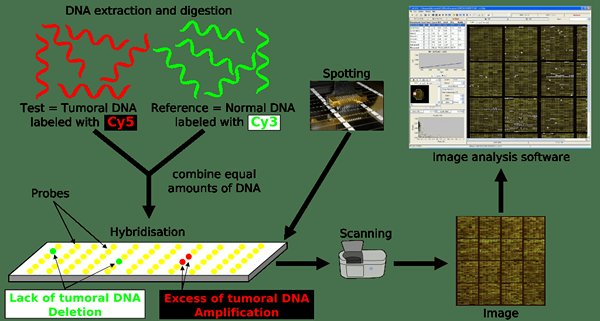
Figura 01: CGH
El procedimiento implica el aislamiento del ADN de dos fuentes: prueba y referencia. Luego, el siguiente paso es el marcado independiente de muestras de ADN de prueba y referencia con fluoróforos de diferentes colores. Más tarde, las dos muestras de ADN se desnaturalizaron para obtener las muestras de ADN monocatenarios. Finalmente, se realizó la hibridación de las dos muestras de ADN resultantes en una relación 1: 1 a una metafase normal de cromosomas. Las diferencias cromosómicas se pueden identificar entre las dos fuentes utilizando un microscopio de fluorescencia y un software de computadora. Además, la técnica CGH solo puede detectar anormalidades cromosómicas desequilibradas.
¿Qué es Array CGH??
Array CGH es un método citogenético molecular totalmente automatizado para analizar la variación del número de copias en el ADN de una muestra de prueba en comparación con una muestra de referencia. Tiene una mayor resolución, y se desarrolló para minimizar las limitaciones de la CGH tradicional. Array CGH combina los principios de CGH con el uso de microarrays. En lugar de usar cromosomas metafase dispersos, este método utiliza diapositivas dispuestas con pequeños segmentos de ADN como objetivos. Estos microarrays se hicieron depositando e inmovilizando una pequeña cantidad de ADN (sondas) en un soporte de vidrio sólido. El tamaño de las sondas puede variar desde oligonucleótidos hasta un cromosoma artificial bacteriano.

Figura 02: Array CGH
Independientemente de las sondas, el método básico de la matriz CGH es similar a CGH. Una vez que se preparan las sondas que contienen microarrays, las dos muestras de ADN (prueba y referencia) marcadas con fluoróforos de diferentes colores se mezclan y se agregan a la microarray. Dado que el ADN en las muestras es monocatenial debido a la desnaturalización, este ADN hibridan con sondas de monedas individuales matriculadas. Usando un sistema de imágenes digitales, es posible identificar las diferencias cromosómicas entre las dos fuentes.
¿Cuáles son las similitudes entre CGH y Array CGH??
- CGH y Array CGH son dos técnicas para detectar variaciones de número de copias.
- Son métodos citogenéticos moleculares.
- Ambos métodos diagnostican anormalidades cromosómicas desequilibradas.
- Los fluoróforos están involucrados en ambos métodos.
- Ambos métodos pueden usarse para detectar los trastornos genéticos debido a las variaciones del número de copias.
- Se usan ampliamente en el análisis genómico comparativo.
¿Cuál es la diferencia entre CGH y Array CGH??
CGH es un método citogenético molecular tradicional para analizar la variación del número de copias en el ADN de una muestra de prueba que tiene una resolución más baja, mientras que la matriz CGH es un método citogenético molecular totalmente automatizado para analizar la variación del número de copias en el ADN de una muestra de prueba que tiene una resolución más alta. Entonces, esta es la diferencia clave entre CGH y Array CGH. Además, en CGH, muestras de ADN de prueba y referencia hibridándose con una metafase normal de cromosomas, pero en la matriz CGH, las muestras de ADN de prueba y de referencia hibridan con las sondas de ADN inmovilizadas que contienen microarray.
La siguiente infografía enumera las diferencias entre CGH y Array CGH en forma tabular para la comparación de lado a lado.
Resumen -CGH vs Array CGH
CGH y Array CGH son dos técnicas citogenéticas moleculares para detectar anormalidades cromosómicas desequilibradas. Se usan ampliamente en el análisis genómico comparativo. CGH es un método citogenético molecular tradicional para analizar la variación del número de copias en el ADN de una muestra de prueba que tiene una resolución más baja. En contraste, Array CGH es un método citogenético molecular totalmente automatizado para analizar la variación del número de copias en el ADN de una muestra de prueba que tiene una resolución más alta. Por lo tanto, esto resume la diferencia entre CGH y Array CGH.
Referencia:
1. "Hibridación genómica comparativa."Una descripción general | Temas de ciencias.
2. Heather, James M., y cadena de Benjamin. "La secuencia de secuenciadores: la historia de la secuenciación del ADN."Genomics, Academic Press, 10 de noviembre. 2015.
Imagen de cortesía:
1. "CGH Schema" de Mbaudis (Talk) (Subidas) - Trabajo propio (CC por 2.5) Vía Commons Wikimedia
2. "Protocolo Array-CGH" de Philippe Hupé-Emmanuel Barillot, Laurence Calzone, Philippe Hupé, Jean-Philippe Vert, Andrei Zinovyev, Biología de sistemas computacionales de Cancer Chapman & “Hall/CRC Matemática” Biología computacional, 2012 (CC BYSA 3.0) a través de Commons Wikimedia