¿Cuál es la diferencia entre Helix-Loop-Helix y Helix-Turn-Helix
El diferencia clave Entre Helix-Loop-Helix y Helix-Turn-Helix es que Helix-Loop-Helix media la dimerización de la proteína, mientras que Helix-Turn-Hélix regula la expresión génica a través de la unión del ADN.
Un motivo de proteína es una secuencia corta y conservada asociada con distintas funciones de ADN. Se asocia principalmente con un sitio estructural especial con una función química o biológica única. Estos motivos contienen pequeñas regiones de estructuras tridimensionales de aminoácidos con diferentes moléculas de proteínas. Por lo general, los motivos individuales contienen solo unos pocos elementos. Helix-Loop-Helix y Helix-Turn-Helix contienen tres elementos. Sus motivos estructurales de proteínas incluyen bucles con longitudes variables y estructuras no especificadas.
CONTENIDO
1. Descripción general y diferencia de claves
2. ¿Qué es Helix-Loop-Helix?
3. ¿Qué es Helix-Turn-Helix?
4. Similitudes-Helix-Loop-Helix y Helix-Turn-Helix
5. Helix-Loop-Helix vs Helix-Turn-Helix en forma tabular
6. Resumen-Helix-Loop-Helix vs Helix-Turn-Helix
¿Qué es Helix-Loop-Helix??
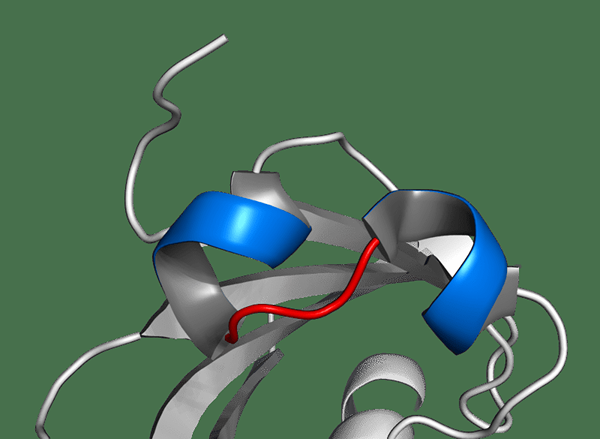
Una hélice helix-loop (HLH) es un motivo estructural de proteínas que define una de las familias más grandes de factores de transcripción dimerizantes. Estos factores de transcripción contienen residuos de aminoácidos para facilitar el mecanismo de unión al ADN, y son diméricos. El motivo estructural de la proteína contiene dos hélices α, y están conectados por un bucle. Una hélice parece más pequeña de las dos hélices, y la flexibilidad del bucle permite la dimerización al empacar y plegar contra otra hélice. La hélice que parece más grande generalmente contiene regiones de unión a ADN. Las proteínas HLH se unen a una secuencia de consenso que se conoce como box e. Una secuencia de consenso es un orden calculado que contiene nucleótidos o residuos de aminoácidos. E-box es un elemento que responde al ADN en algunos eucariotas que actúa como un sitio de unión a proteínas y regula la expresión génica.

Figura 01: motivo Helix-Loop-Helix
Los factores de transcripción HLH son esenciales para el desarrollo y la actividad celular. Las proteínas HLH pertenecen principalmente a seis grupos, que se indican de las letras A a F. Los factores de transcripción incluidos en cada grupo son:
Grupo A: Myod, Myf5, Beta2/Neurod1, SCL, P-Camk, Neurod y Neurogeninas,
Grupo B: Max, C-Myc, N-Myc y TCF4
Grupo C: AHR, BMAL-1-Clock, HIF, NPAS1, NPAS3 y MOP5
Grupo D; EMC
Grupo E: Hey1 y Hey2
Grupo F: EBF1
Dado que la mayoría de los factores de transcripción de HLH son heterodiméricos, la dimerización a menudo los regula.
¿Qué es Helix-Turn-Helix??
Helix-Turn-Helix (HTH) es un motivo estructural de proteína que es capaz de unir el ADN. Cada monómero está organizado con dos hélices α y se une por un cuadro de aminoácidos corto. Esto se une a un surco en la hélice de ADN. Los motivos HTH generalmente regulan la expresión génica. El reconocimiento de HTH y la unión al ADN son realizados por dos hélices α. Una hélice ocupa el extremo N-terminal, mientras que el otro está en C-terminal. En la mayoría de los escenarios, la hélice lleva a cabo el reconocimiento del ADN. Por lo tanto, se conoce como la hélice de reconocimiento. La unión al surco en el ADN tiene lugar a través de una serie de interacciones Van der Waals y enlaces de hidrógeno con bases expuestas. La otra hélice α estabiliza la interacción proteína y de ADN y no juega un papel importante en el reconocimiento. Sin embargo, la hélice de reconocimiento y la hélice restante tienen una orientación similar.
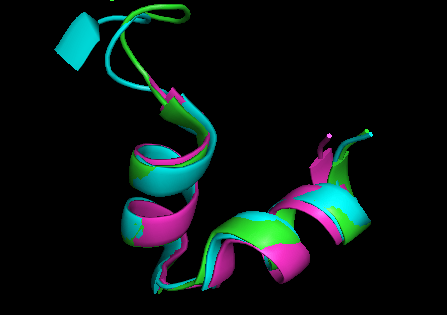
Figura 02: Helix-Turn-Helix de la familia TETR
El HTH se clasifica de acuerdo con la estructura y los arreglos espaciales de las hélices. Los tipos principales son HTH-Hélicos, tri-hélicos, tetra-casicales y alados HTH. El tipo di-hélico es el tipo más simple con dos hélices y un dominio de proteína plegable independiente. El tipo tri-hélico se encuentra en el activador transcripcional myb. Tetra-Helical Type tiene una hélice C-terminal adicional. Finalmente, el HTH alado se forma mediante un paquete de 3 helicos y una hoja beta de 3 o 4 hilos.
¿Cuáles son las similitudes entre Helix-Loop-Helix y Helix-Turn-Helix?
- Helix-Loop-Helix y Helix-Turn-Helix son motivos estructurales de proteínas.
- Ambos contienen un denominador común en factores de transcripción basales y específicos.
- Están presentes en eucariotas.
¿Cuál es la diferencia entre Helix-Loop-Helix y Helix-Turn-Helix?
Helix-loop-hélice media la dimerización de la proteína, mientras que Helix-girn-hélice regula la expresión génica a través de la unión del ADN. Por lo tanto, esta es la diferencia clave entre Helix-Loop-Helix y Helix-Turn-Helix. Además, HLH contiene ciertos proto-oncogenos y genes involucrados en la diferenciación que codifican los factores de transcripción, mientras que HTH contiene muchos genes homeóticos que codifican los factores de transcripción. Además, Helix-Loop-Helix consiste principalmente en hélices alfa unidas por un bucle, mientras que Helix-Turn-Helix consiste principalmente en bucles unidos por un breve soporte de aminoácidos que forma un ritmo.
La siguiente infografía presenta las diferencias entre Helix-Loop-Hélix y Helix-Turn-Helix en forma tabular para la comparación de lado a lado.
Resumen-Helix-Loop-Helix vs Helix-Turn-Helix
Un motivo de proteína es una secuencia corta y conservada asociada con distintas funciones de ADN. Helix-Loop-Helix y Helix-Turn-Helix son dos tipos de motivos estructurales de proteínas. La diferencia clave entre Helix-Loop-Helix y Helix-Turn-Helix es que Helix-Loop-Helix media la dimerización de la proteína, mientras que Helix-Turn-Helix regula la expresión génica a través de la unión al ADN. HLH es un motivo estructural de proteínas que define una de las familias más grandes de factores de transcripción dimerizantes. El motivo estructural de la proteína contiene dos hélices α, y están conectados por un bucle. HTH es un motivo estructural de proteína que es capaz de unir el ADN. Cada monómero está organizado con dos hélices α, y se une por un cuadro corto de aminoácidos y se une a una ranura en la hélice de ADN. Entonces, esto resume la diferencia entre Helix-Loop-Helix y Helix-Turn-Helix.
Referencia:
1. "Helix-loop-hélice básico."Una descripción general | Temas de ciencias.
2. "Helix-Turn-Helix."Una descripción general | Temas de ciencias.
Imagen de cortesía:
1. "Basic Helix Loop Helix" de I, Splette (CC BY-SA 3.0) a través de Commons Wikimedia
2. "HTH alineación de 3 miembros de la familia TETR" por Gabbiochem - Uso de Pymol basado en PDB: 5dy0 depositado por Palanca & Rubio, PDB: 3VIB depositado por Kumaraswami, Shafer, "Brennan y PDB: 3ZQL depositado por Le, Schumacher, Lawson, Brennan "; Buttner (CC por 3.0) a través de Commons Wikimedia