¿Cuál es la diferencia entre MLVA y MLST?
El diferencia clave entre MLVA y MLST es que MLVA utiliza el polimorfismo de las secuencias de ADN repetidas en tándems para caracterizar las especies microbianas, mientras que MLST usa el polimorfismo de las secuencias de ADN de fragmentos internos de múltiples genes de limpieza para caracterizar especies microbianas.
La tipificación microbiana se usa normalmente para determinar la fuente y las rutas de las infecciones. Confirma o descarta brotes. La tipificación microbiana también traza transmisión cruzada de patógenos asociados a la salud y reconoce cepas virulentas. Además, la tipificación microbiana evalúa la efectividad de las medidas de control de las especies microbianas patógenas. MLVA y MLST son dos técnicas biológicas moleculares utilizadas en la tipificación microbiana.
CONTENIDO
1. Descripción general y diferencia de claves
2. Que es mlva
3. Que es mlst
4. Similitudes -MLVA y MLST
5. MLVA vs MLST en forma tabular
6. Resumen -MLVA vs MLST
Que es mlva?
Análisis VNTR de múltiples loci (MLVA) es un método biológico molecular que utiliza el polimorfismo de las secuencias de ADN repetidas tándemamente para caracterizar las especies microbianas. Es un método utilizado para el análisis genético de especies microbianas particulares.
Durante el primer paso de MLVA, cada locus VNTR dirigido se amplifica por PCR con cebadores específicos de la región flanqueante. Luego, los fragmentos obtenidos están separados por electroforesis en un secuenciador capilar. El genotipado de cada locus y los resultados compilados de cada muestra permiten caracterizar un microorganismo cuya especie es conocida. También permite identificar las subespecies a través de la tipificación y comparación de alelos con las bases de datos MLVA.

Figura 01: MLVA
Este método MLVA es más selectivo que el método MLST. Además, el método MLVA no requiere un paso de secuenciación de ADN. Por lo tanto, permite diferenciar la subespecie o las especies clonales.
Que es mlst?
Tipificación de secuencia multilocus (MLST) es una técnica que utiliza el polimorfismo de las secuencias de ADN de fragmentos internos de múltiples genes de limpieza para caracterizar especies microbianas. MLST es una técnica en el análisis genético para la tipificación de múltiples loci.
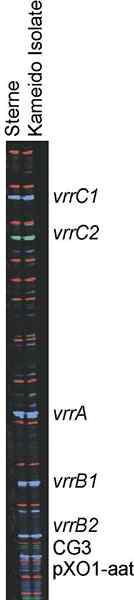
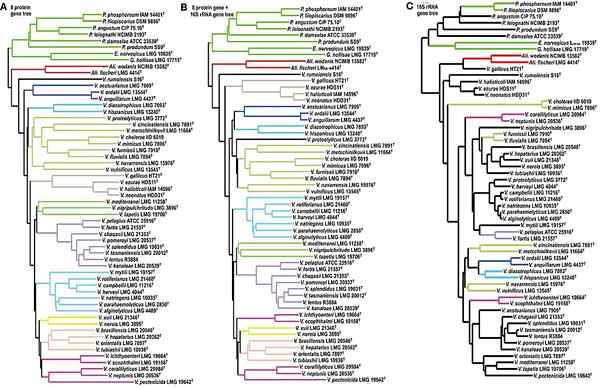
Figura 02: MLST
Es un tipo de tipificación de secuenciación utilizada como técnica de referencia para distinguir diferentes cepas de especies microbianas. Este método se basa en la secuenciación de genes de limpieza. Los genes de limpieza normalmente codifican proteínas esenciales de agentes microbianos como una bacteria. Estas secuencias de genes de limpieza tienen la particularidad de presentar un polimorfismo estable con el tiempo. Por lo tanto, las diferencias en las secuencias de los genes de limpieza son suficientes para distinguir las tensiones entre sí. Además, el primer esquema de MLST que se desarrollaron fue Neisseria meningitidis, El agente causante de la meningitis meningocócica y la septicemia. Desde su introducción, MLST se ha utilizado no solo para los patógenos humanos sino también para los patógenos de las plantas.
¿Cuáles son las similitudes entre MLVA y MLST??
- MLVA y MLST son dos técnicas biológicas moleculares utilizadas en la tipificación microbiana.
- Ambas técnicas pueden usarse para el análisis filogenético microbiano.
- Ambas técnicas se basan en el polimorfismo genético.
- Se pueden usar para la detección de enfermedades humanas que causan patógenos microbianos.
- Ambas técnicas deben ser realizadas por biólogos moleculares calificados.
¿Cuál es la diferencia entre MLVA y MLST??
MLVA es una técnica que utiliza el polimorfismo de las secuencias de ADN repetidas en tándemas para caracterizar las especies microbianas. Mientras tanto, MLST es una técnica que utiliza el polimorfismo de las secuencias de ADN de fragmentos internos de múltiples genes de limpieza para caracterizar especies microbianas. Por lo tanto, esta es la diferencia clave entre MLVA y MLST. Además, el método MLVA es más selectivo que el método MLST.
La siguiente infografía presenta las diferencias entre MLVA y MLST en forma tabular para la comparación de lado a lado.
Resumen -MLVA vs MLST
MLVA y MLST son dos técnicas biológicas moleculares utilizadas en la tipificación microbiana. MLVA utiliza el polimorfismo de las secuencias de ADN repetidas tándemamente para caracterizar las especies microbianas, mientras que MLST usa el polimorfismo de las secuencias de ADN de fragmentos internos de múltiples genes de limpieza para caracterizar las especies microbianas. Entonces, esta es la diferencia clave entre MLVA y MLST.
Referencia:
1. "Análisis de repetición en tándem de número variable de locus múltiple (MLVA)."Centros para el control y la prevención de enfermedades, Centros para el Control y la Prevención de Enfermedades, 16 de febrero. 2016.
2. "Tipo de secuencia multilocus."Una descripción general | Temas de ciencias.
Imagen de cortesía:
1. "1993 48 Kameido aislamientos genotipo" (CC0) a través de Picryl
2. "FMICB-04-00414-G001 (14424086445)" por figuras de filogenia-FMICB-04-00414-G001 (CC por 2.0) a través de Commons Wikimedia